感染组织的宏基因组测序,您选对DNA抽提方法了吗?
本文转自欧易生物
前言
16S核糖体RNA基因(rRNA)的扩增测序及分析是评价临床标本细菌群落多样性常用的方法。该方法利用与16S rDNA高度保守区域结合的引物,通过PCR扩增16S rRNA基因,对扩增得到的产物进行高通量测序及数据分析获得样本中细菌群落的OTU,从而判断样本中的微生物种类。该方法优点是简便快速,缺点是大部分微生物无法鉴定到种的水平。
随着测序平台发展和测序成本的降低,人们对微生物组成的研究从系统发育研究(16S rRNA)转向了宏基因组测序。通过覆盖微生物群落的所有物种的基因组信息,使物种鉴定结果具有更高的特异性和敏感性。宏基因组测序还可以提供酶组成或代谢途径、细菌功能基因组成以及功能与系统发育之间的基因组联系的遗传证据。另外,宏基因组学不仅可以分析来自细菌源的序列,还可以分析来自真菌、病毒和寄生虫的序列。基于上述优势,使得直接分析单个样本中的微生物组数据成为研究临床样本微生物多样性的常用方法。
然而,在研究人类疾病与微生物之间的关系时,由于宿主DNA的污染,宏基因组测序在实验上面临巨大挑战。宿主DNA与微生物DNA的共提取导致后续测序数据中大部分是宿主的基因组数据,从而使微生物组的数据不足,还可能产生干扰下游分析的非特异性信号。解决这个问题有两种方法:要么需要去除宿主DNA,要么需要增加测序深度以获取足够的微生物基因组数据进行后续分析。然而增加测序深度可能更昂贵,因此我们比较了几种去除宿主DNA的微生物基因组抽提方法。
材料与方法
1)材料
糖尿病足感染样本;
2)方法(试剂盒)
实验设计及结果展示
1. 实验设计
糖尿病足感染样本匀浆混样,平均分成15份,每份25mg,用5种方法抽提DNA,并对所得DNA进行定量PCR及16S rDNA 扩增测序以检测物种多样性。

图1
2. 实验结果-DNA质量评价
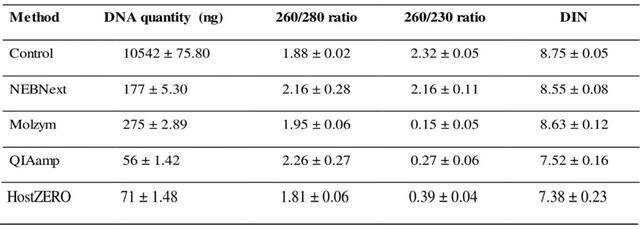
从表1可以看出,五种方法抽提的DNA用NanoDrop™2000c(Thermo Scientific)分光光度法测定提取的DNA产量和纯度约为1.8(260/280),基本符合要求。TapeStation 2200显示DNA完整性(DIN)在7到9之间,这表明DNA没有碎片化(表1)。而Molzym、QIAamp和HostZERO的260/230比值较低。根据Qiagen应用说明,260/230比率低很可能是由于在基于柱的试剂盒中裂解缓冲液经常使用的胍残留所导致,但这不影响下游qPCR检测。
表1 不同方法抽提DNA的各项指标
3. 实验结果-DNA成分鉴定
使用细菌16S特异引物(5’-CCTACGGGAGGCAGCAG-3’和5’-ATTACCGCGGCTGCTGG-3’)和人18S特异引物(5’-GGTGGTGCCCTTCCGTCA-3’ 和 5’-CGATGCGGCGGCGTTATT-3’),通过定量PCR检测DNA样本中宿主和细菌DNA的占比,结果显示,NEBNext法的宿主DNA污染程度高,HostZERO法和QIAamp法的宿主DNA污染程度低(NEBNext>Molzym>QIAamp>HostZERO)(图2)对照样品18S/16S rRNA比值为0.865±0.020。而NEBNext法和Molzym法提取的DNA样本的18S/16S rRNA比值分别为0.701±0.022和0.393±0.057,仍然比较高。
相比之下,QIAamp法和HostZERO法的18S/16S rRNA比值较低,分别为0.027±0.005和0.015±0.007,分别比对照法低了32倍和57倍。
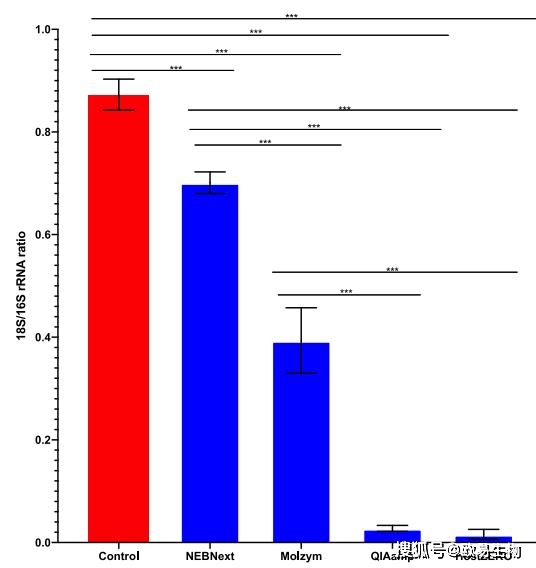
图2
4. 实验结果-微生物DNA占比
通过对定量PCR的数据进行拷贝数计算结果表明,对照样品中细菌DNA含量为(6.7±0.1%)。在去除宿主DNA后,NEBNext法的细菌DNA占比仍然比较低(8.1±0.2%),Molzym法略高(13.6±1.0%)。在QiaAmp法(71.0±2.7%)和HostZero法(79.9±3.1%)中,细菌DNA含量增加了10倍以上(图3)。上述结果表明,HostZERO法和Qiamp法能有效地去除宿主DNA污染,富集微生物DNA。
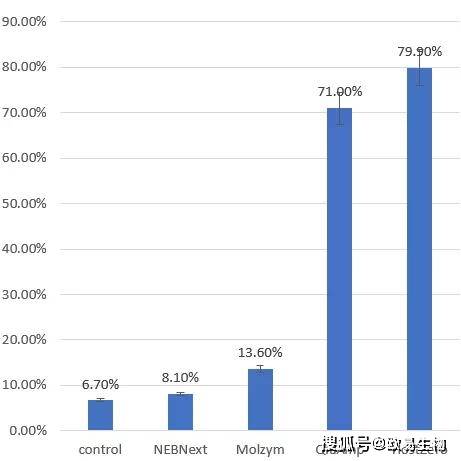
图3
5. 实验结果-16S rRNA扩增测序结果
使用16S rRNA V3V4区引物341(5‘-CCTAYGGGRBGCASCAG-3’)和806(3‘-GGACTACNNGGGTATCTAAT-5’)对样本进行扩增及微生物多样性检测,结果表明:糖尿病足感染组织样本微生物群共包含5个门,这些门在该样本中常见。其中,硬壁门和放线杆菌的成员控制着细菌种群(图4)。NEBNext与对照样品的细菌物种组成(所有五种报告的菌门的提取)相似,这可能是因为这两组样本的DNA提取方法(Roche)一致,NEBNext另外增加了去宿主DNA的步骤。聚类分析也支持这一点,而HostZERO、Molzym和QIAamp形成了一个单独的聚类(图4A和图4B)。在属一级,链球菌主导着糖尿病足感染组织的微生物群,其次是消化链球菌(图4B),与已有报道结果一致。总的来说,本研究中使用的所有微生物组DNA富集方法都成功地识别了糖尿病足感染样本中常见的以及临床上分离出的重要的属,其丰度与先前研究中的报告相对相似。
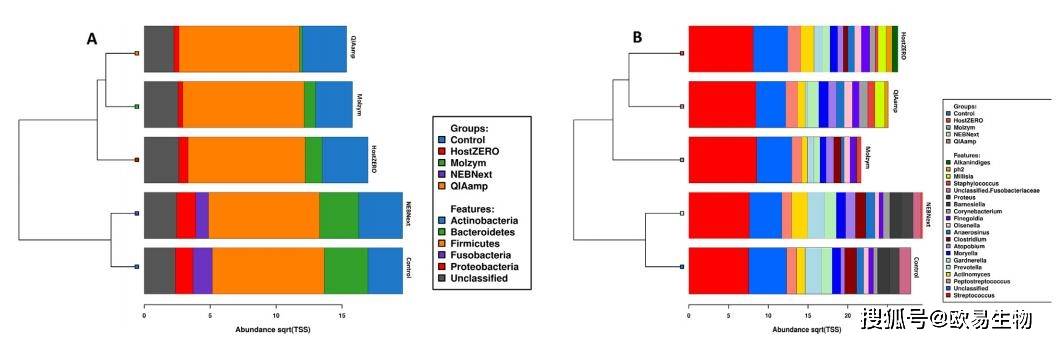
图4
结论
由以上研究结果可知,在测试的五种抽提方法获得的DNA质量相近,但是量的差异比较大。不同的去宿主DNA的方法效果不一,以HostZERO的去宿主效果好,QIAamp的效果其次。用这几种方法抽提的DNA进行16S rDNA扩增对微生物进行检测,结果一致性较高,但是如果用于宏基因组测序,HostZERO和QIAamp抽提方法可能能够更好的去除宿主基因组的影响。
参考文献
Host DNA depletion efficiency of microbiome DNA enrichment methods in infected tissue samples, Fatemah Sadeghpour Heravi, Martha Zakrzewski, Karen Vickery, et. Al., 2020, Journal of Microbiological Methods, https://doi.org/10.1016/j.mimet.2020.105856
附表
表2 几种宿主DNA去除方法的优劣势比较

本文转自欧易生物

